AG Prof. Dr. Oswald
Der Notch Signaltransduktionsweg
Die Entwicklung jedes multizellulären Organismus von der befruchteten Oozyte zu seinem charakteristischen dreidimensionalen Aussehen und seiner Größe ist das Ergebnis komplex koordinierter Genfunktionen. Diese Genfunktionen steuern auf der Ebene einzelner Zellen ein Netzwerk aus Wachstum, Proliferation, Differenzierung, Migration und Apoptose. Die Fähigkeit der Zelle auf externe Signale (z.B. Wachstumsfaktoren und Liganden) durch geeignete Signaltransduktionskaskaden zu reagieren und zu kommunizieren, ist dabei von essentieller Bedeutung. Bei Invertebraten und Vertebraten repräsentiert der Notch Signaltransduktionsweg einen hochkonservierten Mechanismus, der in sehr vielen Entwicklungs- und Differenzierungsprozessen eine entscheidende Rolle spielt. Zusätzlich geht ein dereguliertes Notch Signal oft mit malignen Erkrankungen des Menschen einher.
Die Aufklärung molekularer Mechanismen der Notch-abhängigen Genregulation ist ein Schwerpunkt der Arbeitsgruppe und trägt einerseits zum grundlegenden Verständnis von Transkription bei. Andererseits ergeben sich daraus Möglichkeiten zur molekularen Einflußnahme (z. B. durch „small molecules“) für neue Therapiekonzepte.
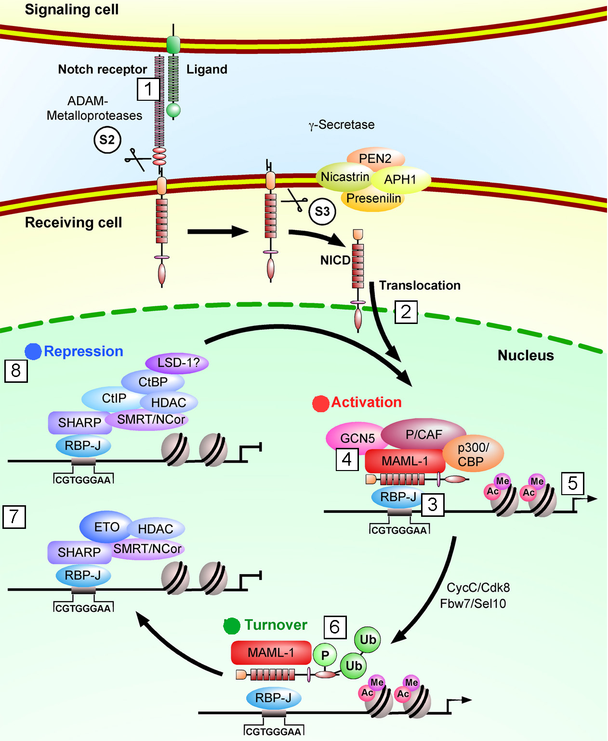
Notch Rezeptoren und ihre Liganden sind membranständige Proteine (1) eines Signaltransduktionswegs, der in vielen Entwicklungs- und Differenzierungsprozessen eine entscheidende Rolle spielt. Ligandbindung induziert verschiedene proteolytische Spaltungsprozesse am Notch Rezeptor (S2 und S3) bei denen ADAM Metalloproteasen und ein Gamma-Sekretase Komplex beteiligt sind. Dies führt zur nukleären Translokation der intrazellulären Domäne (NICD) (2). NICD bindet selbst keine DNA, sondern interagiert mit dem sequenzspezifisch DNA-bindenden Protein RBP-Jkappa (RBP-J) (3), um nach Rekrutierung eines Koaktivator Komplexes (4) die Transkription von Notch-Zielgenen zu aktivieren (5). Die Abschaltung der Transkriptionsaktivierung erfolgt über eine schnelle Degradation von NICD im Zellkern nach spezifischen postranslationalen Modifikationen [Phosphorylierung (P), Ubiquitinylierung (Ub)] (6) In Säugern interagieren die intrazellulären Domänen aller vier Notch Rezeptoren mit RBP-J. Somit kommt diesem Protein eine entscheidende Bedeutung im Notch Signaltransduktionsweg zu. RBP-J ist ein hochkonserviertes, ubiquitär exprimiertes Protein und fungiert, solange der Notch Signalweg inaktiv ist, als transkriptioneller Repressor. Seine transkriptionsreprimierende Wirkung entfaltet RBP-J durch die Rekrutierung von Multiprotein-Korepressor-Komplexen (7 und 8). Spezifische Histon-Deacetylasen (HDACs) scheinen dabei wichtige Bestandteile zu sein. Als neue Korepessoren in diesem Komplex konnten von uns SHARP, CtBP und ETO identifiziert werden. SHARP bindet einerseits direkt an RBP-J und rekrutiert andererseits HDACs und weitere HDAC assoziierte Proteine. Der genaue molekulare Mechanismus der RBP-Jkappa vermittelten transkriptionellen Repression ist jedoch noch wenig verstanden. Durch proteinbiochemische, molekularbiologische, zellbiologische und entwicklungs-biologische Verfahren werden in unserer Arbeitsgruppe die molekularen Mechanismen der RBP-J abhängigen Repression, sowie die der NICD vermittelten Transaktivierung näher untersucht (Abb. 1).
Arbeiten der letzten Jahre haben gezeigt, dass während Notch abhängiger Differenzierungsprozesse eine komplexe Modulation der Notch Aktivität stattfindet. Diese Modulation geschieht vor allem auf der Ebene der Notch-Liganden aber auch am Notch-Rezeptor selbst durch spezifische posttranslationale Modifikationen. Wir konnten ein neues, bisher nicht charakterisiertes Phosphoprotein, RITA, identifizieren das über einen unerwarteten nukleo-zytoplasmatischen Transportmechanismus die Verfügbarkeit von RBP-J für aktives Notch im Zellkern und somit die Notch Aktivität negativ reguliert. Im Krallenfrosch (Xenopus laevis) als Modellsystem ist RITA für die neuronale Differenzierung essentiell und wirkt der durch aktives Notch induzierten Hemmung der neuronalen Differenzierung entgegen. Weiterhin induziert RITA die neuronale Differenzierung im Zellkultur Modell. Somit stellt RITA einen evolutionär hochkonservierten Modulator des Notch Signalwegs dar, der zumindest bei neuronalen Differenzierungsprozessen eine kritische Rolle einnimmt. Die Aufklärung der Signaltransduktionskaskade(n), die eine Funktion von RITA als negativer Modulator von Notch beeinflussen, sollen in Zukunft biochemisch, zellbiologisch und mit geeigneten Mausmodellen durchgeführt werden.
Innerhalb von DFG (SFB1074, www.uni-ulm.de/einrichtungen/sfb-1074.html) und BMBF (Molecular Systems Biology of impaired Stem Cell Function and Regeneration during Aging “SyStaR”, www.uni-ulm.de/in/systar.html) finanzierten Projekten werden in der Arbeitsgruppe Veränderungen im Notch Signalweg bei Tumoren und zellulären Alterungsprozessen in verschiedenen Tiermodellen und beim Menschen untersucht.

Neben der Aufklärung molekularer Mechanismen der Notch/RBP-J vermittelten Transkriptionsregulation ist ein weiterer Forschungsschwerpunkt der Arbeitsgruppe die Isolierung, Charakterisierung und Optimierung neuer fluoreszierender Proteine aus nichtbiolumineszenten Nesseltieren sowie die Entwicklung geeigneter Biosensoren für die Anwendung in lebenden Organismen und Zellen (Abb. 2).
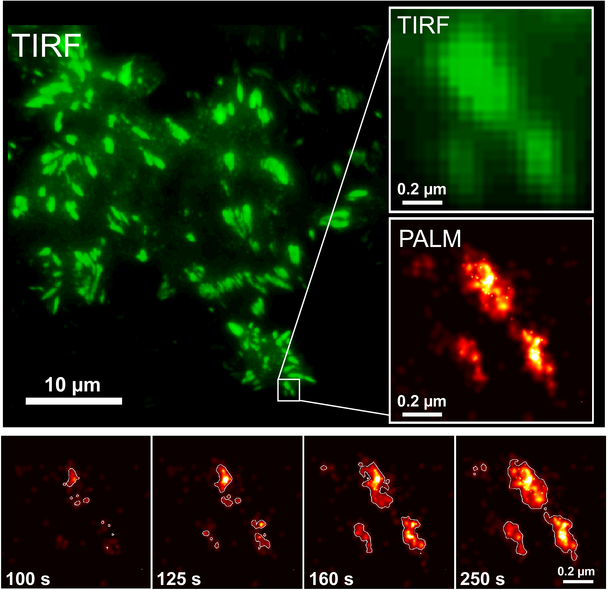
Aus den Arbeiten dieses Forschungsschwerpunktes sind u. a. das tiefrot-fluoreszierende Protein eqFP611, dessen monomere Variante mRuby sowie die von grün nach rot fotokonvertierbaren Proteine EosFP und mCavRFP hervorgegangen. In verschiedenen Arbeiten konnten wir die Anwendbarkeit dieser Proteine als molekulare Marker, sowohl für entwicklungsbiologische Fragestellungen als auch in eukaryontischen Zellsystemen aufzeigen und dort maßgebliche Beiträge leisten. Kürzlich konnten wir das von mEosFP abgeleitete mIrisFP für die Anwendung in der optischen Höchstauflösungs-Mikroskopie vorstellen. Die Fluoreszenzfarbe von mIrisFP lässt sich, wie bei mEosFP, durch Bestrahlung mit Laserlicht von grün nach rot umschalten. Darüber hinaus kann sowohl die grüne als auch die rote Form gezielt ein- und ausgeschaltet werden. Durch diese Eigenschaften ist es nun möglich, Proteinwanderungen in der lebenden Zelle in optischer Höchstauflösung zu beobachten (Abb. 3).
Team
Leiter
Mitarbeiter
Wissenschaftliche Publikationen
Publikationen Prof. Dr. Franz Oswald
Originalarbeiten (Auswahl):
- Agrawal, M., Schwarz, P., Giaimo, B. D., Bedzhov, I., Corbacioglu, A., Weber, D., Gaidzik, V.I., Jahn, N., Rucker, F.G., Schroeder, T., Kindler, T., Wattad, M., Gotze, K., Lubbert, M., Salwender, H., Ringhoffer, M., Lange, E., Koller, E., Thol, F., Heuser, M., Ganser, A., Bullinger, L., Paschka, P., Döhner, H., Geiger, H., Borggrefe, T., Döhner, K. & Oswald, F. (2020) Functional and clinical characterization of the alternatively spliced isoform AML1-ETO9a in adult patients with translocation t(8;21)(q22;q22.1) acute myeloid leukemia (AML). Leukemia 34 (2) 630-634.
- Yuan, Z., VanderWielen, B. D., Giaimo, B. D., Pan, L., Collins, C. E., Turkiewicz, A., Hein, K., Oswald, F., Borggrefe, T. & Kovall, R. A. (2019) Structural and Functional Studies of the RBPJ-SHARP Complex Reveal a Conserved Corepressor Binding Site. Cell Rep 26: 845-854 e846
- Close, V., Close, W., Kugler, S. J., Reichenzeller, M., Yosifov, D. Y., Bloehdorn, J., Pan, L., Tausch, E., Westhoff, M. A., Döhner, H., Stilgenbauer, S., Oswald, F*. & Mertens, D*. (2019) FBXW7 mutations reduce binding of NOTCH1, leading to cleaved NOTCH1 accumulation and target gene activation in CLL. Blood 133: 830-839 (*corresponding authors)
- Hurtado, C., Safarova, A., Smith, M., Chung, R., Bruyneel, A. A. N., Gomez-Galeno, J., Oswald, F., Larson, C. J., Cashman, J. R., Ruiz-Lozano, P., Janiak, P., Suzuki, T. & Mercola, M. (2019) Disruption of NOTCH signaling by a small molecule inhibitor of the transcription factor RBPJ. Sci Rep 9: 10811
- Kindermann, B., Valkova, C., Kramer, A., Perner, B., Engelmann, C., Behrendt, L., Kritsch, D., Jungnickel, B., Kehlenbach, R. H., Oswald, F., Englert, C. & Kaether, C. (2019) The nuclear pore proteins Nup88/214 and T-ALL-associated NUP214 fusion proteins regulate Notch signaling. J Biol Chem 294 (31): 11741-11750.
- Wolf, D., Smylla, T. K., Reichmuth, J., Hoffmeister, P., Kober, L., Zimmermann, M., Turkiewicz, A., Borggrefe, T., Nagel, A. C., Oswald, F., Preiss, A. & Maier, D. (2019) Nucleo-cytoplasmic shuttling of Drosophila Hairless/Su(H) heterodimer as a means of regulating Notch dependent transcription. Biochim Biophys Acta Mol Cell Res 1866: 1520-1532.
- Kreis, N. N., Steinhäuser, K., Ritter, A., Klöble, P., Hoock, S. C., Roth, S., Louwen, F., Oswald, F. & Yuan, J. (2019) Potential involvement of RITA in the activation of Aurora A at spindle poles during mitosis. Oncogene 38: 4199-4214.
- Giaimo, B. D., Ferrante, F., Vallejo, D. M., Hein, K., Gutierrez-Perez, I., Nist, A., Stiewe, T., Mittler, G., Herold, S., Zimmermann, T., Bartkuhn, M., Schwarz, P., Oswald, F., Dominguez, M. & Borggrefe, T. (2018) Histone variant H2A.Z deposition and acetylation directs the canonical Notch signaling response. Nucleic Acids Res 46: 8197-8215.
- Tharehalli, U., Svinarenko, M., Kraus, J. M., Kühlwein, S., Szekely, R., Kiesle, U., Scheffold, A., Barth, T. F. E., Seufferlein, T., Oswald, F., Katz, S-F. & Lechel A (2018) YAP Activation Drives Liver Regeneration after Cholestatic Damage Induced by Rbpj Deletion. Int J Mol Sci 19: doi:10.3390.
- Berger, A. W., Raedler, K., Langner, C., Ludwig, L., Dikopoulos, N., Becker, K. F., Slotta-Huspenina, J., Quante, M., Schwerdel, D., Perkhofer, L., Kleger, A., Zizer, E., Oswald, F., Seufferlein, T. & Meining, A. (2018) Genetic Biopsy for Prediction of Surveillance Intervals after Endoscopic Resection of Colonic Polyps: Results of the GENESIS Study. United European Gastroenterol J 6: 290-299.
- Oswald, F., Klöble, P., Ruland, A., Rosenkranz, D., Hinz, B., Butter, F., Ramljak, S., Zechner, U., Herlyn, H. (2017) The FOXP2-Driven Network in Developmental Disorders and Neurodegeneration. Front Cell Neurosci 11: doi: 10.3389/fncel.2017.00212. eCollection 02017.
- Steinhäuser, K., Klöble, P., Kreis, N. N., Ritter, A., Friemel, A., Roth, S., Reichel, J. M., Michaelis, J., Rieger, M. A., Louwen, F., Oswald, F. & Yuan, J. (2017) Deficiency of RITA results in multiple mitotic defects by affecting microtubule dynamics. Oncogene 36: 2146-2159.
- Tabaja, N., Yuan, Z., Oswald, F. & Kovall, R. A. (2017) Structure-function analysis of RBP-J-interacting and tubulin-associated (RITA) reveals regions critical for repression of Notch target genes. J Biol Chem 292: 10549-10563.
- Thiel, V. N., Giaimo, B. D., Schwarz, P., Soller, K., Vas, V., Bartkuhn, M., Blätte, T. J., Döhner, K., Bullinger, L., Borggrefe, T., Geiger, H. & Oswald, F. (2017) Heterodimerization of AML1/ETO with CBFbeta is required for leukemogenesis but not for myeloproliferation. Leukemia 31: 2491-2502.
- Oswald, F*., Rodriguez, P., Giaimo, B. D., Antonello, Z. A., Mira, L., Mittler, G., Thiel, V. N., Collins, K. J., Tabaja, N., Cizelsky, W., Rothe, M., Kühl, S. J., Kühl, M., Ferrante, F., Hein, K., Kovall, R. A., Dominguez, M. & Borggrefe, T. (2016) A phospho-dependent mechanism involving NCoR and KMT2D controls a permissive chromatin state at Notch target genes. Nucleic Acids Res 44: 4703-4720 (*corresponding author).
- Vegi, N. M., Klappacher, J., Oswald, F., Mulaw, M. A., Mandoli, A., Thiel, V. N., Bamezai, S., Feder, K., Martens, J. H., Rawat, V. P., Mandal, T., Quintanilla-Martinez, L., Spiekermann, K., Hiddemann, W., Döhner, K., Döhner, H., Stunnenberg, H. G., Feuring-Buske, M. & Buske, C. (2016) MEIS2 Is an Oncogenic Partner in AML1-ETO-Positive AML. Cell Rep 16: 498-507
- Hein, K., Mittler, G., Cizelsky, W., Kühl, M., Ferrante, F., Liefke, R., Berger, I. M., Just, S., Sträng, J. E., Kestler, H. A., Oswald, F*. & Borggrefe, T*. (2015) Site-specific methylation of Notch1 controls the amplitude and duration of the Notch1 response. Sci. Signal. 8: ra30 (*corresponding author)
- Gittins, J. R., D'Angelo, C., Oswald, F., Edwards, R. J. & Wiedenmann, J. (2015). Fluorescent protein-mediated colour polymorphism in reef corals: multicopy genes extend the adaptation/acclimatization potential to variable light environments. Mol Ecol., 24, 453-465.
- Jung, C., Mittler, G., Oswald, F. & Borggrefe, T. (2013). RNA helicase Ddx5 and the noncoding RNA SRA act as coactivators in the Notch signaling pathway. Biochim Biophys Acta. 1833, 1180-1189.
- Schmeisser, M. J., Baumann, B., Johannsen, S., Vindedal, G. F., Jensen, V., Hvalby, O. C., Sprengel, R., Seither, J., Maqbool, A., Magnutzki, A., Lattke, M., Oswald, F., Boeckers, T. M. & Wirth, T. (2012) IkappaB Kinase/Nuclear Factor kappaB-Dependent Insulin-Like Growth Factor 2 (Igf2) Expression Regulates Synapse Formation and Spine Maturation via Igf2 Receptor Signaling. J Neurosci, 32, 5688-5703
- Wacker, S. A., Alvarado, C., von Wichert, G., Knippschild, U., Wiedenmann, J., Clauß, K., Nienhaus, G. U., Hameister, H., Baumann, B., Borggrefe, T., Knöchel, W. & Oswald, F. (2011). RITA, a novel modulator of Notch signaling, acts via nuclear export of RBP-J Embo J. 30, 43-56.
- Fuchs, J.*, Böhme, S.*, Oswald, F.*, Hedde, P. N., Krause, M., Wiedenmann, J. & Nienhaus, G. U. (2010). A photoactivatable marker protein for pulse-chase imaging with superresolution. Nat Methods 7, 627-630. (*eq cont)
- Liefke, R.*, Oswald, F.*, Alvarado, C., Ferres-Marco, D., Rodriguez, P., Dominguez, M. & Borggrefe, T. (2010). Histone demethylase KDM5A is an integral part of the core Notch-RBP-J repressor complex. Genes Dev. 24, 590-601, (*eq cont).
- Brosi, H., Reiser, M., Rajasalu, T., Spyrantis, A., Oswald, F., Boehm, B. O., & Reinhold Schirmbeck (2009) Processing in the Endoplasmic Reticulum Generates an Epitope on the Insulin A Chain that Stimulates Diabetogenic CD8 T Cell Responses. J. Immunology, 183, 187-195.
- Hedde, P. N., Fuchs, J., Oswald, F., Wiedenmann, J. & Nienhaus, G. U. (2009) Online image analysis software for photoactivation localization microscopy. Nat. Methods 10, 689-690.
- Kredel, S., Oswald, F., Nienhaus, K., Deuschle, K., Röcker, C., Wolff, M., Heilker, R., Nienhaus, G. U. & Wiedenmann, J. (2009) mRuby, a bright monomeric red fluorescent protein for labeling of subcellular structures PLoS ONE. 4, e4391.
- Salat, D., Liefke, R., Wiedenmann, J., Borggrefe, T. & Oswald, F. (2008) ETO, but not leukemogenic fusion protein AML1/ETO, augments RBP-Jkappa/SHARP-mediated repression of Notch target genes. Mol Cell Biol. 28, 3502-3512.
- Kredel, S*., Nienhaus, K*., Oswald, F*., Wolff, M., Ivanchenko, S., Cymer, F., Jeromin, F., Spindler, K. D., Heilker, R., Nienhaus, G. U. & Wiedenmann, J. (2008) Optimized and Far-red Emitting Variants of Fluorescent Protein eqFP611. Chem Biol. 15, 224-233, (*eq. cont.)
- Wacker, S. A.*, Oswald, F*., Wiedenmann, J. & Knöchel, W. (2007) A green to red photoconvertible protein as an analyzing tool for early vertebrate development. Dev Dyn, 236, 473-480. (*eq. cont.).
- Oswald, F., Schmitt, F., Leutenegger, A., Ivanchenko, S., D'Angelo, C., Salih, A., Maslakova, S., Bulina, M., Schirmbeck, R., Nienhaus, G. U., Matz, M. V. & Wiedenmann, J. (2007) Contributions of host and symbiont pigments to the coloration of reef corals. Febs J, 274, 1102-1109.
- Oswald, F., Winkler, M., Cao, Y., Astrahantseff, K., Bourteele, S., Knöchel, W. & Borggrefe, T. (2005) RBP-Jkappa/SHARP recruits CtIP/CtBP corepressors to silence Notch target genes. Mol Cell Biol, 25, 10379-10390.
- Wiedenmann, J*., Ivanchenko, S*., Oswald, F*., Schmitt, F., Röcker, C., Salih, A., Spindler, K. D. & Nienhaus, G. U. (2004) EosFP, a flourescent marker protein with UV-inducible green-to-red fluorescence conversion. Proc. Natl. Acad. Sci. U S A, 101, 15905-15910. (*eq. cont).
- Oswald, F., Kostezka, U., Astrahantseff, K., Bourteele, S., Dillinger, K., Zechner, U., Ludwig, L., Wilda, M., Hameister, H., Knöchel, W., Liptay, S. & Schmid, R. M. (2002) SHARP is a novel component of the Notch/RBP-Jkappa signalling pathway. Embo J., 21, 5417-5426.
Reviews:
- Oswald, F*. & Kovall, R. A*. (2018) CSL-Associated Corepressor and Coactivator Complexes. Adv Exp Med Biol 1066: 279-295 (*corresponding authors).
- Giaimo, B. D., Oswald, F. & Borggrefe, T. (2017) Dynamic chromatin regulation at Notch target genes. Transcription 8: 61-66.
- Borggrefe, T., Lauth, M., Zwijsen, A., Huylebroeck, D., Oswald, F. & Giaimo, B. D. (2016) The Notch intracellular domain integrates signals from Wnt, Hedgehog, TGFbeta/BMP and hypoxia pathways. Biochim Biophys Acta 1863: 303-313.
- Borggrefe, T. & Oswald, F. (2016) Setting the Stage for Notch: The Drosophila Su(H)-Hairless Repressor Complex. PLoS Biol 14: e1002524.
- Borggrefe, T. & Oswald, F. (2014). Keeping notch target genes off: a CSL corepressor caught in the act. Structure, 22, 3-5.
- Wiedenmann, J., Gayda, S., Adam, V., Oswald, F., Nienhaus, K., Bourgeois, D. & Nienhaus, G. U. (2011). From EosFP to mIrisFP: structure-based development of advanced photoactivatable marker proteins of the GFP-family. J. Biophotonics 4 (6) 377-390.
- Borggrefe, T. & Oswald, F. (2009) The Notch signaling pathway: Transcriptional regulation at Notch target genes. Cell Mol Life Sci. 66, 1631-1646
- Wiedenmann, J., Oswald, F. & Nienhaus, G. U. (2009) Fluorescent proteins for live cell imaging: opportunities, limitations, and challenges. IUBMB Life. 61 (11) 1029-1042.
